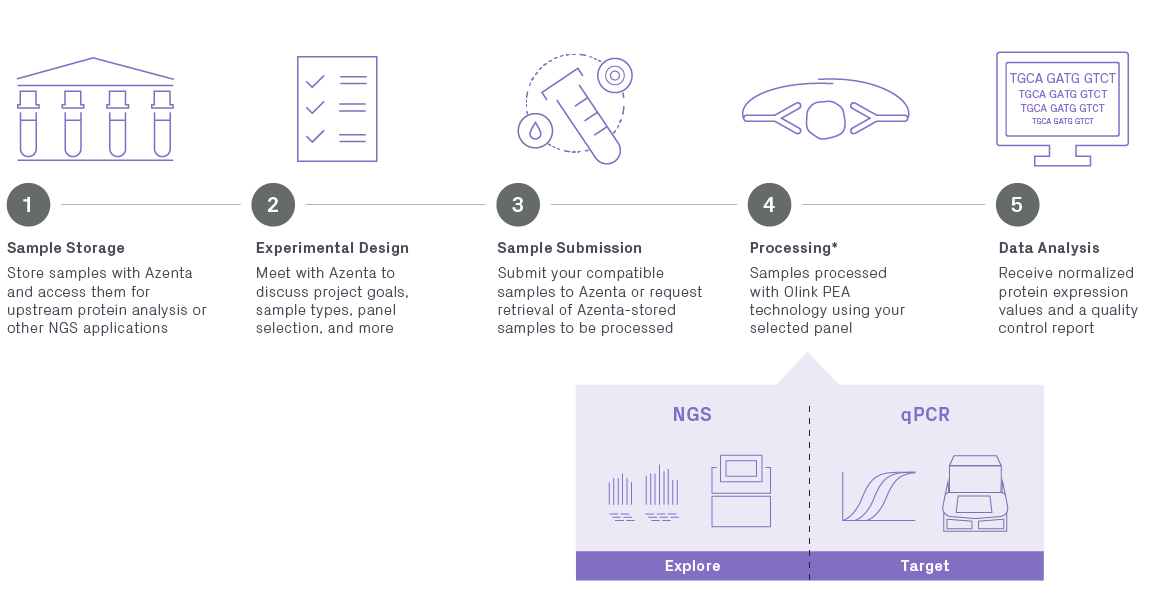
Olink Proteomics によるタンパク質バイオマーカーの検出
Olink Proteomics®のPEA法(Proximity Extension Assay)を活用したジーンウィズのタンパク質バイオマーカー検出サービスでは、血中タンパク質の検出と定量化を実現することにより、疾患や薬剤応答の詳細な生物学的知見を提供します。従来の方法と異なり、このアプローチでは、高い特異性と感度により、タンパク質を高スループットで同時解析することが可能です。しかも、使用するサンプル量は最小限ですみます。Olink Proteomics社認証サービスプロバイダーとして、多検体処理のExplorer HT、中規模のTarget 48/96、カスタマイズしたFlex/Focusまで、多様なプロジェクトに対応します。このサービスは研究用途(RUO)だけでなく、CAP:米国病理学会による認定とCLIA:臨床検査室改善法の認証を取得している当社の認証施設でもお引き受けすることが可能です。
このプロテオミクスサービスをアゼンタの ゲノム や トランスクリプトーム ソリューションと統合した、マルチオミクスによるアプローチにより、治験での開発、疾患、薬剤応答に寄与する分子変化の包括的な理解が実現します。

タンパク質バイオマーカー発見のためのOlinkパネル
| Explore HT | Olink Explore 3072/384 | Olink Target 96 | Olink Target 48 | Flex & Focus |
| ハイスループット プロテオミクスのための次世代ソリューションです。 Olink Explore HT には、特異性が証明された 5,400 以上のタンパク質が含まれており、トップレベルの経路を 100% カバーしています。 | <ヒト疾患にフォーカスした384-plexの8つのパネルで構成されているOlink Exploreは、柔軟性とスケーラビリティの両方を兼ね備えたパネルです。ライブラリ全体を解析することも、また、384-plexパネルのうち一つまたは複数を選択することも可能です。 | 特定領域のヒト疾患や主要な生物学的プロセスを念頭に、慎重な設計によって構築した15のパネルから選択することができます。マウスモデルを用いて研究している研究者の方向けに、Target 96 Mouse Exploratoryも提供しています。 | 現在市販されている最も範囲の広いヒトサイトカイン炎症パネルの一つであるOlink Target 48では、炎症プロセスや疾患研究に最も関連性の深いタンパク質の分析を実施し、治験の裏付けとすることが可能です | オーダーメイドのバイオマーカーパネル製品です。Olink Flex および Olink Focus を使用すると、 1 つのバイオマーカー パネルあたり最大 21 個のヒトタンパク質のターゲットを選択して組み合わせることができます。 |
| これまでで最も包括的なパネルであり、2,536 個の新しいバイオマーカーが含まれています。 わずか 2 µL のサンプルをインプットとして 5,300 以上のタンパク質を測定できます。データは、相対定量値(NPX)で得られます。 |
これらのパネルは次の4つの疾患領域に体系化されています。:
|
疾患領域:
|
Target 48のサイトカインパネルで解明できるのは:
|
Olink の広範なバイオマーカー ライブラリから事前検証済みのアッセイを選択して組み合わせます。このアッセイは 99% の組み合わせ可能性を持ち、標準的な Olink パネルに関連する品質、性能、特異性、および感度を維持します。絶対定量 (pg/mL) または相対定量 (NPX) でデータを得ます。 |
| アッセイリストのダウンロード: Explore HT Protein Panels > |
アッセイリストのダウンロード: Explore 3072/384 Protein Panels > |
アッセイリストのダウンロード: Target 96 Protein Panels > |
アッセイリストのダウンロード: Target 48 Protein Panels > |
Click to download the assay list: Flex/Focus Protein Panels > |
用途
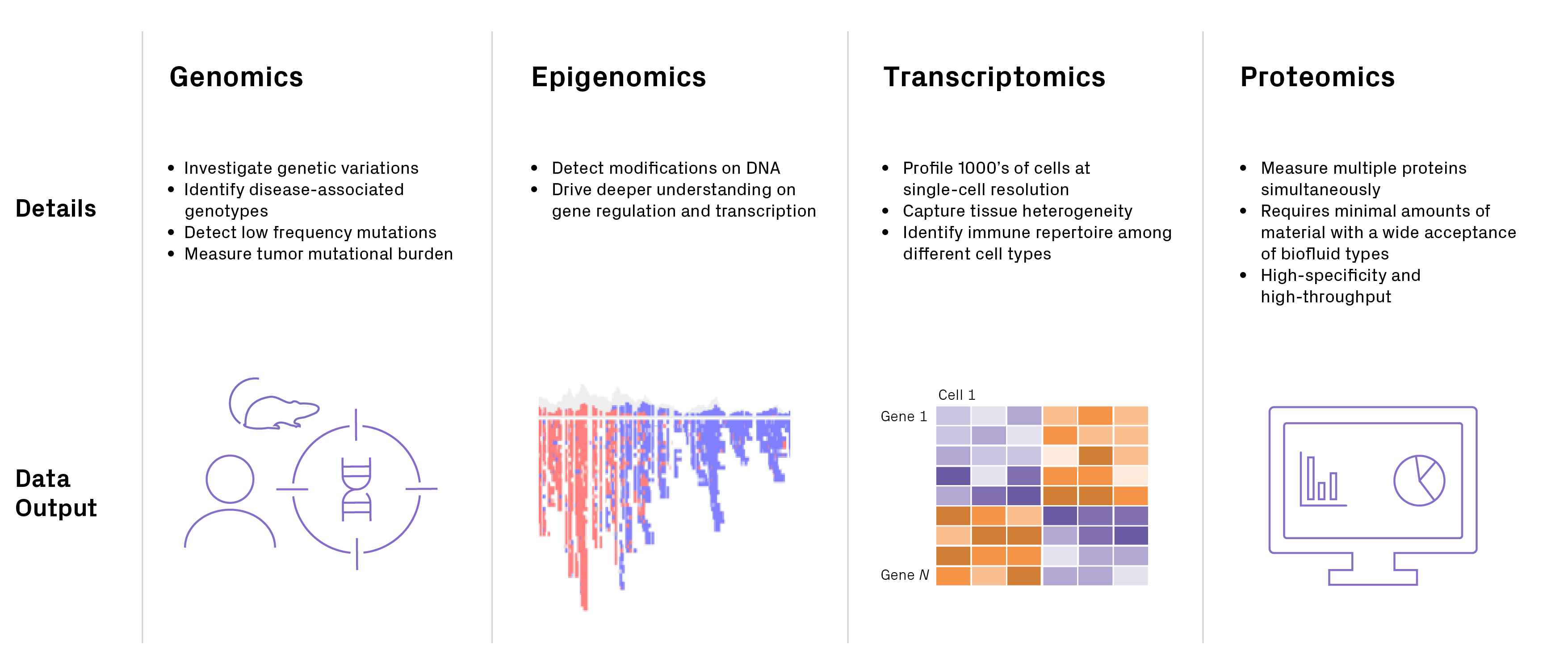
バイオマーカー探索のためのマルチオミクスアプローチ
ゲノミクス、トランスクリプトミクス、エピゲノミクス、プロテオミクスはそれぞれ、ヒト細胞の内部の働きについてのユニークで重要な知見を提供します。これらのマルチオミクス ツールを統合すると、関係性とパターンに関する詳細なデータが得られ、創薬ターゲットの探索を加速および拡大するのに役立ちます。
ジーンウィズの強み
-
安心のサンプル管理
ストレージソリューションのノウハウを生かして -
迅速な納期
解析サイトにサンプルが到着してから最速10営業日で納品 -
信頼できるパートナー
Olink認証プロバイダです。北米トップ20の製薬会社がアゼンタを選択
Blog │ The Power of Proteomics
The landscape of drug development is constantly evolving, fueled by groundbreaking scientific advancements. While genetics has played a central role in understanding diseases, proteomics provides a more comprehensive picture of disease phenotypes and mechanisms. In this blog, discover how researchers and clinicians are using Olink proteomics to reshape the outcomes of their clinical studies.
Blog | Comparing Mass Spectrometry, ELISA, and Olink for Proteomics
The field of proteomics has evolved significantly in the last decade, with several technologies now available to assay protein biomarkers in clinical studies. In this article, we compare three popular approaches for proteomics – mass spectrometry, ELISA, and the Olink® Proximity Extension Assay (PEA) – and highlight their strengths and limitations.
White Paper │ Multiplex Analysis of Inflammatory Proteins: A Comparative Study Across Multiple Platforms
Multiplexing technologies are often challenged by cross-reactive binding or interference contributing to the signal readout, which can lead to poor specificity. Olink®‘s proprietary PEA technology circumvents these challenges by use of a dual recognition approach with matched pairs of antibodies labeled with complimentary DNA oligos. This white paper verifies the quality of Olink technology relative to two common multiplex proteomics platforms widely used in protein biomarker research.
Tech Note | Proteomic Biomarker Discovery to Support Clinical Trials
Traditionally preclinical exploratory research and clinical trial biomarker detection and measurement have been restricted to a few indicators measured by mass spectrometry and ELISA. Discover how GENEWIZ has implemented fully validated protein biomarker discovery services using the Olink® Proximity Extension Assay (PEA) technology capable of detecting and measuring hundreds to thousands of proteins simultaneously.
Tech Note | High-Plex Exploratory Biomarker Discovery to Support Proteomics and Multiomics Research
The development of novel strategies for mid-to high-plex protein detection and quantification enables greater utilization of protein biomarkers across the drug discovery and development process. Using Olink® Proximity Extension Assay (PEA) technology, GENEWIZ has deployed fully validated protein biomarker discovery services for up to 1 million data points per experiment with comparable sensitivity to traditional methods and utilization of lower sample amounts.
Blog | How Olink technology is revolutionizing proteomics and multiomics
Current proteomics technologies often present limitations in terms of throughput, specificity, and cost. However, new innovations, such as Olink Proteomics’ PEA technology, are overcoming these challenges by enabling hundreds to thousands of proteins to be measured in a single sample. In this blog, discover how GENEWIZ is leveraging PEA technology for protein biomarker discovery, its applications, and how it can complement genomics data to accelerate drug development.
Webinar | Precision Proteomics: How to Enable Biomarker Identification in Health and Disease
Hear directly from Olink Proteomics about precision protein analysis as a tool to improve health outcomes for patients with cancer, inflammatory, or age-related diseases. Take this chance to discover how you can bring your protein biomarker discovery to the next level with Olink PEA technology.
Webinar | Do More with Less: Harnessing the Power of Multiomics from a Single Sample
The -omics era has greatly expanded the repertoire of approaches available for researchers and clinicians to unravel the complexities underpinning human health. In this recording from the GENEWIZ from Azenta Breakfast Talk at ASHG 2022, Dr. Andrea O’Hara presents a workflow to rapidly produce a diverse set of multiomics results, including genomics, epigenomics, transcriptomics, and proteomics, from a single blood draw.
Poster | A Demonstrated Workflow to Unleash the Multiomic Potential of a Single Blood Draw
The multiomics era has greatly expanded the repertoire of approaches available for researchers and clinicians to unravel the complexity underpinning human health. Explore how the single blood draw multiomics workflow from GENEWIZ can drive more significant insights and innovation in human health applications.